MATLAB-Alternativen in Python

Eines vorne weg: Es gibt kein Python-Programm, das MATLAB eins zu eins ersetzen könnte.
Allerdings hat Python an sich einige Ähnlichkeiten zu MATLAB. So sind beides Interpreter-Sprachen.
Auch können beide wahlweise in einer interaktiven Konsole (z.B. ipython) verwendet werden oder
als Skripte (.m-Files/.py-Files) ausgeführt werden. Und mit NumPy exisitiert ein mächtiges Python-Modul,
dass eine Fülle von mathematischen Funktionen zur Verfügung stellt.
Python-Packages
Hinweis: Alle Beispiele wurden für Python 2.7 geschrieben.
| Python-Package | Vergleichbare Matlab-Toolbox/ Funktionalität | Website |
| NumPy | Grundpaket für das Rechnen u.a. mit Vektoren und Matrizen in Python | NumPy |
| SciPy | Zusammenfassung mehrerer Python-Pakete für Mathematik, Naturwissenschaften und Technik | SciPy |
| matplotlib | Umfangreiches Python-Modul zum Erzeugen von 2D- und 3D-Graphen | matplotlib |
| SymPy | Symbolic Toolbox | SymPy |
| IPython | Interaktive Python-Shell mit einigen Zusatzfunktionen | IPython |
| Pandas | Datenanalyse, Statistik | Pandas |
| AudioLazy | Digital Signal Processing (DSP) Package | AudioLazy |
| PYO | Python-Modul für digital signal processing | PYO |
| PIL | Python Image Library | PIL |
| MayaVi | Tool zum Visualisieren von Daten in 3D-Graphen | MayaVi |
| NeuroLab | Neural Network Toolbox | NeuroLab |
| OMPC | Wandelt Matlab-.m-Files in Pythoncode um oder führt sie innerhalb eines Python-Skripts aus. (Funktionsumfang noch stark eingeschränkt) | OMPC |
Code-Beispiele
Die folgenden Beispiel geben nur einen Bruchteil der Möglichkeiten wieder. Weitere Anwendungen finden sich in den Dokumentationen des jeweiligen Pakets.
Sinn der Beispiele ist es, einige Anregungen für die Ver- und Anwendung der verschiedenen Python-Module zugeben. Die hier dargestellten Probleme sind dafür nur
mittel zum Zweck. Es wurde dafür auf Problemstellungen zurück gegriffen, denen man so oder so ähnlich auch in einem Ingenieurstudiums begegnet. Erklärungen zu den
Problemstellungen sollen nur zum Code-Verständnis beitragen.
IPython
Die interaktive Pythonkonsole wird mit dem Kommando 'ipython' in der Konsole gestartet. Hier kann man nun ganz normalen Pythoncode eingeben.
Die Reaktion auf die Eingabe erfolgt sofort nach betätigen der Eingabetaste.
C:\Users\Sven>ipython
Python 2.7.3 (default, Apr 10 2012, 23:31:26) [MSC v.1500 32 bit (Intel)]
Type "copyright", "credits" or "license" for more information.
IPython 0.13.1 -- An enhanced Interactive Python.
? -> Introduction and overview of IPython's features.
%quickref -> Quick reference.
help -> Python's own help system.
object? -> Details about 'object', use 'object??' for extra details.
In [1]: import numpy as np
In [2]: for i in range(10):
...: print i
...:
0
1
2
3
4
5
6
7
8
9
In [3]: %save example 1-2
Das Ergebnis lässt sich bei bedarf am Ende mit dem Kommando '%save DATEINAME' als Python-Skript speichern.
Dabei lässt sich angeben, welche Zeilennummern gespeichert werden sollen.
Ein nützliches Werkzeug um einige Zeilen Code zusammen mit Anmerkungen zu speichern ist IPython Notebook.
Gestartet wird Notebook über die Kommandozeile.
Microsoft Windows [Version 6.1.7601]
Copyright (c) 2009 Microsoft Corporation. Alle Rechte vorbehalten.
C:\Users\Sven>ipython notebook
[NotebookApp] Using existing profile dir: u'C:\\Users\\Sven\\.ipython\\profile_d
efault'
[NotebookApp] Serving notebooks from C:\Users\Sven
[NotebookApp] The IPython Notebook is running at: http://127.0.0.1:8888/
[NotebookApp] Use Control-C to stop this server and shut down all kernels.
Nach der Eingabe öffnet sich Notebook im Browser.
Fängt man mit der Python-Programmierung an, ist es empfehlenswert sich Python(x,y) herunterzuladen und zu installieren.
Dabei wird nicht nur der Python-Interpreter, sondern auch eine Reihe nützlicher Python-Pakete (z.B. Numpy, Ipython und Matplotlib) installiert.
Außerdem ist auch Spyder, eine IDE, enthalten.
NumPy, SciPy und Matplotlib
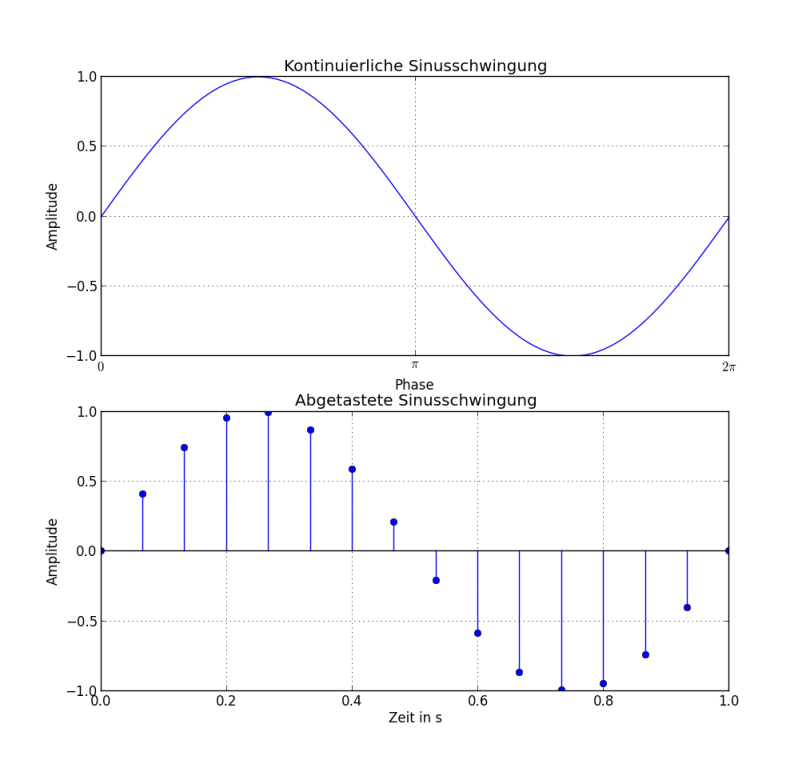
Als Beispiel für den Einstieg in NumPy und matplotlib dient eine einfache Sinusschwingung.
Anhand des Beispiels wird das Erstellen eines Vektors in NumPy sowie verschiedene Darstellungsmöglichkeiten in matplotlib gezeigt.
y_{(t)}=y_{0}\cdot sin(\omega t+\varphi _{0})
NumPy bietet verschiedene Funktionen zum Umgang mit Vektoren (NumPy-Arrays) an.
# -*- coding: utf-8 -*-
"""
Created on Thu May 15 19:05:51 2014
@author: Sven
"""
import matplotlib.pyplot as plt
import numpy as np
from pylab import *
y0 = 1.0 # Amplitude
freq = 1.0 # Frequenz in Hz
tau = 1.0/freq # Periodendauer in s
f_abtast = 15.0 # Abtastfrequenz in Hz
phi = 0 # Phasenwinkel
x1 = np.arange(0, tau+0.01, 0.01) # Vektor x-Achse
y1 = y0 * np.sin(2*np.pi*freq*x1+phi) # Vektor y-Achse
x2 = np.arange(0, tau+(tau/f_abtast), tau/f_abtast)
y2 = y0 * np.sin(2*np.pi*freq*x2+phi) # Vektor y-Achse
labels =['$0$','$\pi$','$2\pi$'] # Beschriftung x-Achse
# Kontinuierliches Signal plotten
ax = plt.subplot(2, 1, 1) # Subplot anlegen
plt.plot(x1, y1, '-') # Daten als kontinuierliche Linie plotten
plt.xticks(x1, labels) # Achsenbeschriftung durch den Inhalt von labels ersetzen
ax.xaxis.set_ticks(np.arange(0, 1.01, 0.5)) # Achsenskalierung einstellen
plt.title('Kontinuierliche Sinusschwingung') # Graph-Title
plt.grid() # Gitter anzeigen
plt.xlabel('Phase') # Benennung x-Achse
plt.ylabel('Amplitude') # Bennenung y-Achse
# Zeit- und wertdiskretes Signal plotten
plt.subplot(2, 1, 2)
markerline, stemlines, baseline = plt.stem(x2, y2, 'b-') # Daten in zeit- und wertdiskreter Darstellung
plt.setp(markerline, 'markerfacecolor', 'b') # Linien formatieren
plt.setp(stemlines, 'markerfacecolor', 'b')
plt.setp(baseline, 'color','black', 'linewidth', 1)
plt.title('Abgetastete Sinusschwingung')
plt.xlabel('Zeit in s')
plt.ylabel('Amplitude')
plt.grid()
plt.show() # Graph anzeigen
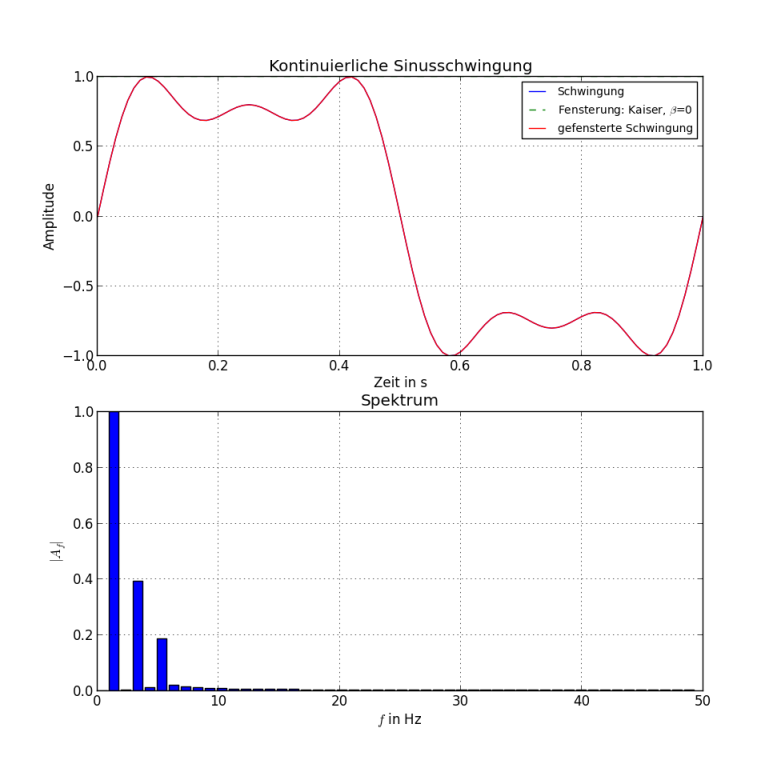
Als zweites NumPy-Beispiel wird von einem Sinussignal mit Oberschwingungen eine Fouriertransformation erstellt und geplottet.
# -*- coding: utf-8 -*-
"""
Created on Thu May 15 19:05:51 2014
@author: Sven
"""
import matplotlib.pyplot as plt
import numpy as np
from pylab import *
y1 = 1.0 # Amplitude Grundschwingung bzw. 1. Harmonische
y3 = 0.4 # Amplitude 3. Harmonische
y5 = 0.2 # Amplitude 5. Harmonische
freq = 1.0 # Frequenz in Hz
tau = 1.0/freq # Periodendauer in s
time_step = 0.01 # Abtastfrequenz
phi = 0 # Phasenwinkel
x = np.arange(0, tau+time_step, time_step) # Vektor x-Achse
harm1 = y1 * np.sin(2*np.pi*freq*x+phi) # Vektor 1. Harmonische
harm3 = y3 * np.sin(2*np.pi*3*freq*x+phi) # Vektor 3. Harmonische
harm5 = y5 * np.sin(2*np.pi*5*freq*x+phi) # Vektor 5. Harmonische
y = np.add(np.add(harm1, harm3), harm5) # Vektoren addieren
# Spektrumsanalyse
n = len(y)
freq_ax = np.arange(n)/(n*time_step)
freq_ax = freq_ax[range(n/2)]
#window_func = np.hanning(n) # Vektor Fenster-Funktion
#window_func = np.blackman(n) # Vektor Fenster-Funktion
#window_func = np.hamming(n) # Vektor Fenster-Funktion
window_func = np.kaiser(n,0) # Vektor Fenster-Funktion
y_window = np.multiply(window_func, y) # Fensterfunktion mit Schwingung multiplizieren
Y = np.abs(np.fft.fft(y_window)/n) # FFT berechnen und Betrag bilden
Y = np.multiply(2, Y[range(n/2)]) # Spiegelung des Spektrums verwerfen und dafuer Amplituden verdoppeln
plt.subplot(2, 1, 1) # Subplot anlegen
plt.plot(x, y, '-', label='Schwingung') # Daten als kontinuierliche Linie plotten
plt.plot(x, window_func, '--', label=r'Fensterung: Kaiser, $\beta$=0') # Fenster-Funktion plotten
plt.plot(x, y_window, '-', label='gefensterte Schwingung') # Fenster-Funktion plotten
plt.title('Kontinuierliche Sinusschwingung') # Graph-Titel
plt.grid() # Gitter anzeigen
plt.xlabel('Zeit in s') # Benennung x-Achse
plt.ylabel('Amplitude') # Bennenung y-Achse
plt.legend(loc='best', prop={'size':10}) # Legende zufuegen
plt.subplot(2, 1, 2) # Subplot anlegen
plt.bar(freq_ax, Y) # Daten als Balkendiagramm plotten
plt.title('Spektrum')
plt.xlabel('$f$ in Hz')
plt.ylabel('$|A_{f}|$')
plt.grid()
plt.show()
In NumPy gibt es auch verschiedene Fenster-Funktionen. Diesen wird die Vektorlänge n übergeben und man bekommt einen Vektor mit der Fensterfunktion zurück.
Mittels elementweisen Multiplizieren kann damit dann das Signal gefenstert werden. Die im Plot verwendete Kaiser-Fensterung mit β=0 entspricht einem Rechteck-Fenster.
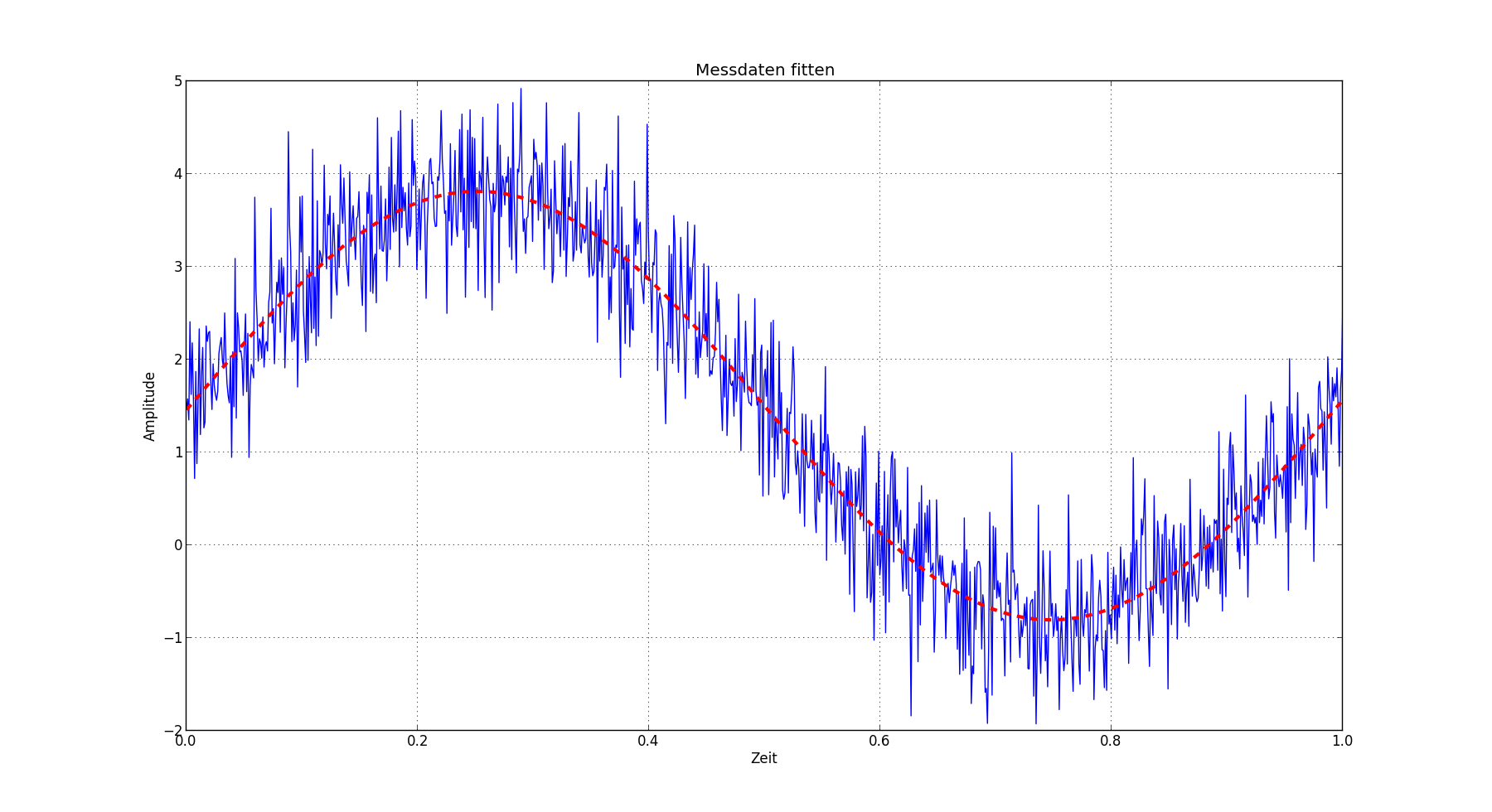
Häufig will man eine Menge an Messpunkten durch eine Funktion annähern. In diesem Beispiel soll eine Sinusfunktion in ein verrauschtes Messsignal gefittet werden.
Hierzu kann man die "curve_fit"-Funktion des optimize-Moduls von SciPy verwendet werden. Dieser Funktion werden die Messdaten, sowie die Zielfunktion (als Python-Funktion formuliert) übergeben.
Als Returnwerte erhält man zwei Arrays, wobei das erste Array die Parameter der gefitteten Funktion enthält. Die Lösung wird mittels der Methode der kleinsten Fehlerquadrate ermittelt.
# -*- coding: utf-8 -*-
"""
Created on Sun Mar 20 14:13:43 2016
@author: Sven
"""
import matplotlib.pyplot as plt
import numpy as np
from scipy.optimize import curve_fit
def sinus_func(x, freq, phi, amp, offset):
'''Sinusfunktion mit den vier zu optimierenden Groessen'''
return (amp * np.sin(2*np.pi*freq*x+phi)) + offset
x = np.linspace(0, (1.0/1.0), 1000) # Vektor x-Achse
y = sinus_func(x, 1.0, 0, 2.3, 1.5) # Datenpunkte
y = np.add(y, 0.5*np.random.randn(len(x))) # Verrauschte Daten
ax = plt.subplot(1, 1, 1) # Subplot anlegen
plt.plot(x, y, 'b-') # Daten plotten
popt, pcov = curve_fit(sinus_func, x, y) # Kurve fitten
y_fit = sinus_func(x, popt[0], popt[1], popt[2], popt[3])
plt.plot(x, y_fit, 'r--', linewidth=3.0) # Daten plotten
plt.title('Messdaten fitten') # Graph-Title
plt.grid() # Gitter anzeigen
plt.xlabel('Zeit') # Benennung x-Achse
plt.ylabel('Amplitude') # Bennenung y-Achse
plt.show() # Graph anzeigen
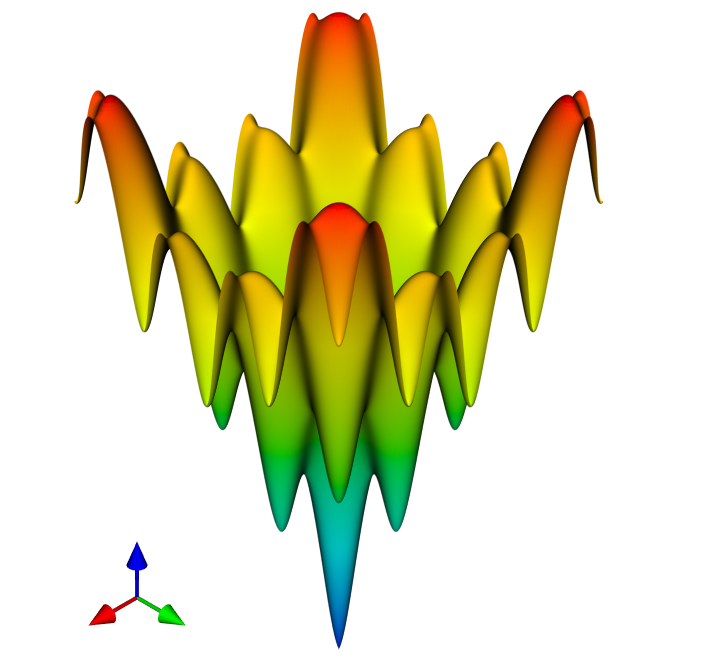
Wir bleiben für das nächste Beispiel beim Thema Optimierung. Anhand der Ackley-Funktion soll gezeigt werden, wie man mit matplotlib Daten in dreidimensionalen Graphen darstellt.
Außerdem wird in diesem Beispiel das Rechnen mit NumPy-Vektoren vertieft.
f_{(x,y)}=-20\cdot e^{-0,2\sqrt{\frac{x^{2}+y^{2}}{2}}}-e^{\frac{cos(2\pi x)+cos(2\pi y)}{2}}+20+e
# -*- coding: utf-8 -*-
"""
Created on Fri Nov 14 13:51:41 2014
@author: Sven
"""
import numpy as np
from matplotlib import pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
import time
def montecarlo(linksunten, rechtsoben, n, func):
'''Sucht Ackley-Funktion nach Minimum ab mittels Monte-Carlo-Verfahren'''
# Variablen für aktuelles Minimum
min_y = 0
min_x = 0
min_z = 0
# Suchgebiet
xu = linksunten[0]
yu = linksunten[1]
xo = rechtsoben[0]
yo = rechtsoben[1]
d_x = xo-xu
d_y = yo-yu
# Laufzeiterfassung
start = time.clock()
# Zufälligen xy-Koordinate im Suchgebiet erzeugen
point = [xu + d_x*np.random.rand(), yu + d_y*np.random.rand()]
min_x,min_y = np.meshgrid([point[0]],[point[1]])
# z-Koordinate berechnen
min_z = func(min_x,min_y)[0][0]
for i in range(n-1):
point = [xu + d_x*np.random.rand(), yu + d_y*np.random.rand()]
px,py = np.meshgrid([point[0]],[point[1]])
pz = func(px,py)[0][0]
# Testen ob ein neues Minimum gefunden wurde
if pz < min_z:
min_z = pz
min_x = px
min_y = py
print "Laufzeit in s: ",time.clock()-start
return min_x[0][0],min_y[0][0],min_z
def ackley(x, y):
'''Ackley'''
a = -20 * np.exp(-0.2*np.sqrt((x**2 + y**2)/2))
b = -np.exp((np.cos(2*np.pi*x) + np.cos(2*np.pi*y))/2)
z = a + b + 20 + np.exp(1)
return z
# Testprogramm mit Ausgabe in Matplotlib
x = np.arange(-2.0, 2.0, 0.01)
y = np.arange(-2.0, 2.0, 0.01)
X,Y = np.meshgrid(x, y)
Z = ackley(X, Y)
# Minimum suchen
px,py,pz = montecarlo([-2.0,-2.0],[2.0,2.0],1000, func=ackley)
print "Minimum bei: ",px,',',py,',',pz
# Plotten
fig = plt.figure(figsize=(10, 7))
ax = fig.gca(projection='3d')
surf = ax.plot_wireframe(X, Y, Z, rstride=10, cstride=10, linewidth=0.5)
min_point = ax.scatter(px,py,pz,c=u'r')
ax.set_title("Ackley-Funktion")
ax.set_xlabel("x")
ax.set_ylabel("y")
ax.set_zlabel("z")
plt.show()
Im Code wird zur Veranschaulichung eine Ackley-Funktion geplottet und anschließen mit einem Monte-Carlo-Verfahren das Minimum der Funktion gesucht.
Beim Monte-Carlo-Verfahren werden einige zufällige Punkte erzeugt, um dann die Funktion an diesen Punkten auszuwerten.
Punkte die ein niedrigeres Ergebnis lieferen, als die vorhergehenden werden gespeichert, während die alten Punkte verworfen werden.
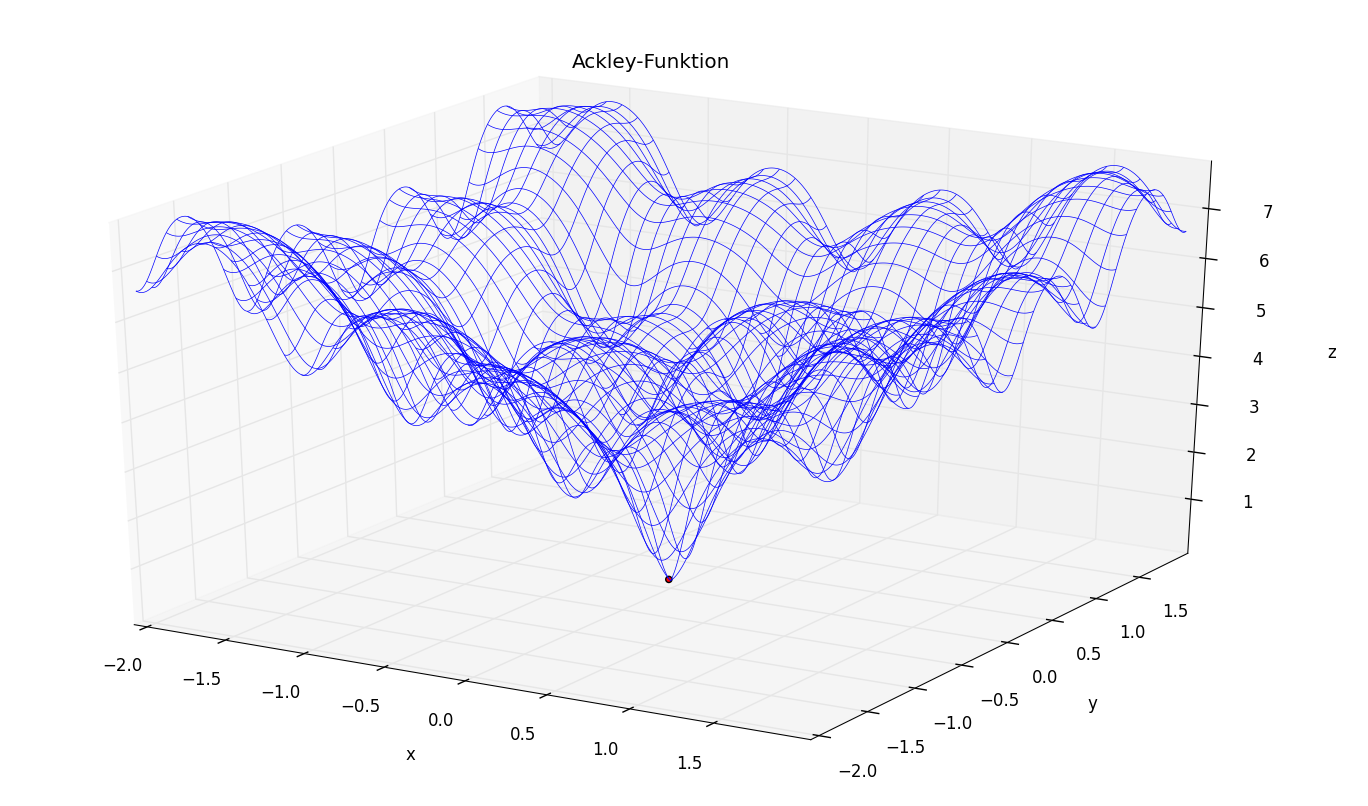
Das gefundene Minimum wird durch einen roten Punkt markiert. Die Qualität des gefundenen Punkts wächst mit der Anzahl der Testpunkte.
Diese lässt sich im Programm über den Paramter n variieren. Auch ist es hilfreich, wenn man die ungefähre Lage des Minimums kennt und so das Suchgebiet
zu Beginn eingrenzen kann. Das Suchgebiet wird dabei über durch ein Rechteck begrenzt, von welchem die linke, untere und rechte, obere Ecke angegeben werden.
Da das Verfahren das Suchgebiet zufällig absucht, hat man keine Garantie, dass die gefundene Lösung tatsächlich das Minimum darstellt.
Als Verbesserung bietet sich darum eine Kombination mit einem systematischen Suchverfahren an. Mit Monte-Carlo-Suche mit wenigen Iterationen wird das Gebiet
grob abgesucht, um dann das gefundene vermeintliche Minimum als Ausgangspunkt für eine systematische Suche, beispielsweise mit dem Gradienten-Verfahren, nach dem tatsächliche globale Minimum zu nutzen.
Ein weiteres Beispiel zum Rechnen mit Vektoren und Matrizen in NumPy stammt aus dem Bereich der Objekterkennung.
Um Objekte anhand bestimmter Merkmale leichter ihrer Klasse zuordnen zu können, ist es manchmal hilfreich eine Koordinatentransformation durchzuführen.
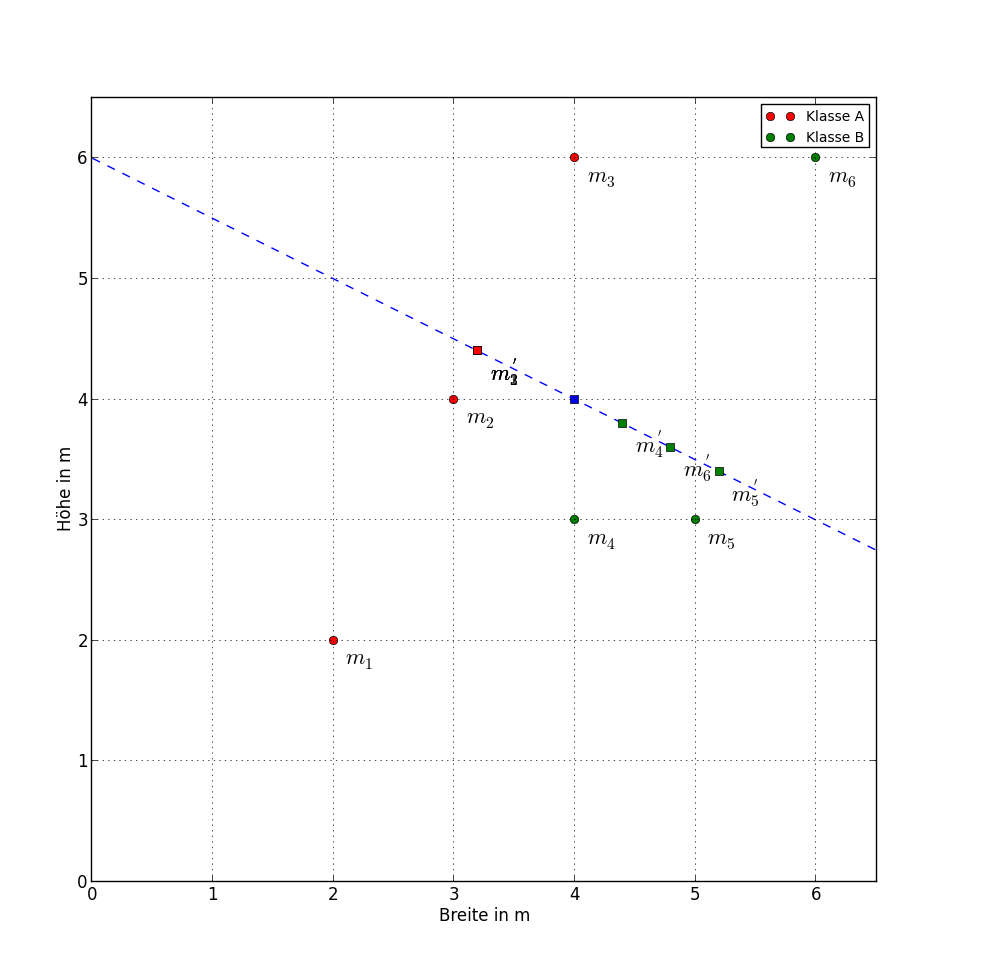
Eine mögliche Anwendung wäre es beispielsweise, anhand von Höhe und Breite (Merkmale) zu entscheiden, ob es sich bei einem Fahrzeug (Objekt) um einen LKW oder einen PKW (Klasse) handelt.
\text{Klasse A: }m_{1}=\begin{pmatrix}
2\\
2
\end{pmatrix}\text{; }
m_{2}=\begin{pmatrix}
3\\
4
\end{pmatrix}\text{; }
m_{3}=\begin{pmatrix}
4\\
6
\end{pmatrix}
\text{Klasse B: }m_{4}=\begin{pmatrix}
4\\
3
\end{pmatrix}\text{; }
m_{5}=\begin{pmatrix}
5\\
3
\end{pmatrix}\text{; }
m_{6}=\begin{pmatrix}
6\\
6
\end{pmatrix}
Trägt man die Punkte der beiden Beispielklassen in ein Koordinatensystem ein, merkt man schnell dass diese sich nicht so einfach trennen lassen.
# -*- coding: utf-8 -*-
"""
Created on Fri Nov 14 13:52:18 2014
@author: Sven
"""
import numpy as np
import matplotlib.pyplot as plt
import sympy as sym
def transform(point, Aufpunkt, Richtungsvektor):
s = sym.Symbol('s')
runningPoint = Aufpunkt + Richtungsvektor*s
# Skalarprodukt ist Null wenn beide Vektoren senkrecht aufeinanderstehen
s = sym.solve(np.dot(runningPoint-point,Richtungsvektor),s)
return Aufpunkt + Richtungsvektor*s
# Klasse A
m1 = np.array([2.0,2.0])
m2 = np.array([3.0,4.0])
m3 = np.array([4.0,6.0])
# Klasse B
m4 = np.array([4.0,3.0])
m5 = np.array([5.0,3.0])
m6 = np.array([6.0,6.0])
m_A = (m1+m2+m3)*(1.0/3.0) # Mittelwert Klasse A
m_B = (m4+m5+m6)*(1.0/3.0) # Mittelwert Klasse B
m_0 = (1.0/6.0)*(m1+m2+m3+m4+m5+m6)
#Streumatrizen
s1 = np.multiply( ( m1 - m_A ),np.transpose(np.matrix( m1 - m_A ))) + \
np.multiply(( m2 - m_A ),np.transpose(np.matrix( m2 - m_A ))) + \
np.multiply(( m3 - m_A ),np.transpose(np.matrix( m3 - m_A )))
s2 = np.multiply( ( m4 - m_B ),np.transpose(np.matrix( m4 - m_B ))) + \
np.multiply(( m5 - m_B ),np.transpose(np.matrix( m5 - m_B ))) + \
np.multiply(( m6 - m_B ),np.transpose(np.matrix( m6 - m_B )))
s_w = s1 + s2
w = np.dot(np.linalg.inv(s_w),(m_A - m_B))
# Matrix in Vektor umwandeln
w = np.squeeze(np.asarray(w))
font = {'family' : 'serif',
'color' : 'black',
'weight' : 'normal',
'size' : 16,
}
# Graph erzeugen
fig = plt.figure()
# equal setzt das Seitenverhältnis auf 1:1
ax = fig.add_subplot(111, aspect='equal')
# Klassenmittelwert plotten
p_m0 = ax.plot(m_0[0], m_0[1], 'bs')
# Objekte der Klasse A plotten...
p_m1 = ax.plot(m1[0], m1[1], 'ro', label='Klasse A')
p_m2 = ax.plot(m2[0], m2[1], 'ro')
p_m3 = ax.plot(m3[0], m3[1], 'ro')
# ...und beschriften
ax.text(m1[0]+0.1, m1[1]-0.2, r'$m_{1}$', fontdict=font)
ax.text(m2[0]+0.1, m2[1]-0.2, r'$m_{2}$', fontdict=font)
ax.text(m3[0]+0.1, m3[1]-0.2, r'$m_{3}$', fontdict=font)
# Objekte der Klasse B plotten...
p_m4 = ax.plot(m4[0], m4[1], 'go', label='Klasse B')
p_m5 = ax.plot(m5[0], m5[1], 'go')
p_m6 = ax.plot(m6[0], m6[1], 'go')
# ...und beschriften
ax.text(m4[0]+0.1, m4[1]-0.2, r'$m_{4}$', fontdict=font)
ax.text(m5[0]+0.1, m5[1]-0.2, r'$m_{5}$', fontdict=font)
ax.text(m6[0]+0.1, m6[1]-0.2, r'$m_{6}$', fontdict=font)
# Achse für transformierte Merkmale
p1 = m_0+w
p2 = m_0-w
line = plt.plot([p1[0],p2[0]],[p1[1],p2[1]], 'b--')
# Punkte auf transformierte Merkmalsachse projizieren
m1_transf = transform(m1, m_0, w)
m2_transf = transform(m2, m_0, w)
m3_transf = transform(m3, m_0, w)
ax.plot(m1_transf[0], m1_transf[1], 'rs')
ax.plot(m2_transf[0], m2_transf[1], 'rs')
ax.plot(m3_transf[0], m3_transf[1], 'rs')
ax.text(m1_transf[0]+0.1, m1_transf[1]-0.2, r"$m_{1}^{'}$", fontdict=font)
ax.text(m2_transf[0]+0.1, m2_transf[1]-0.2, r"$m_{2}^{'}$", fontdict=font)
ax.text(m3_transf[0]+0.1, m3_transf[1]-0.2, r"$m_{3}^{'}$", fontdict=font)
m4_transf = transform(m4, m_0, w)
m5_transf = transform(m5, m_0, w)
m6_transf = transform(m6, m_0, w)
ax.text(m4_transf[0]+0.1, m4_transf[1]-0.2, r"$m_{4}^{'}$", fontdict=font)
ax.text(m5_transf[0]+0.1, m5_transf[1]-0.2, r"$m_{5}^{'}$", fontdict=font)
ax.text(m6_transf[0]+0.1, m6_transf[1]-0.2, r"$m_{6}^{'}$", fontdict=font)
ax.plot(m4_transf[0], m4_transf[1], 'gs')
ax.plot(m5_transf[0], m5_transf[1], 'gs')
ax.plot(m6_transf[0], m6_transf[1], 'gs')
ax.axis([0, 6.5, 0, 6.5])
ax.grid()
plt.xlabel(u'Breite in m')
plt.ylabel(u'Höhe in m')
ax.legend(loc='best', prop={'size':10})
plt.show()
SymPy
OMPC
MayaVi
MayaVi ist speziell zum Visualisieren von dreidimensionalen Daten gedacht. Als kleines Beispiel zum Einstieg soll wieder die Ackley-Funktion geplottet werden.
Um MayaVi in Spyder verwenden zu können, muss man eine Änderung an den Einstellungen vornehmen, indem man unter
Tools->Preferences->Console->External Modules->Enthought Tool Suite->ETS_TOOLKIT Qt4 in wx ändert.
# -*- coding: utf-8 -*-
"""
Created on Mon Dec 29 13:23:48 2014
@author: Sven
"""
from mayavi import mlab
import numpy as np
def ackley(x, y):
a = -20 * np.exp(-0.2*np.sqrt((x**2 + y**2)/2))
b = -np.exp((np.cos(2*np.pi*x) + np.cos(2*np.pi*y))/2)
z = a + b + 20 + np.exp(1)
return z
x = np.arange(-2.0, 2.0, 0.01)
y = np.arange(-2.0, 2.0, 0.01)
X,Y = np.meshgrid(x,y)
Z = ackley(X,Y)
s = mlab.mesh(X,Y,Z)
mlab.show()
PIL - Python Image Library
 Rocking Wombat
Rocking Wombat





